Der Studiengang Prozessmanagement und Business Intelligence (PMBI) der Fachhochschule Oberösterreich am Campus Steyr arbeitet seit 2008 an Forschungsprojekten zu medizinischen Analysen und Benchmarking mit verschiedenen österreichischen Krankenhausträgern und Behörden. 2015 wurden diese Aktivitäten mit dem wissenschaftlichen Benchmarking-Programm für Krankenhäuser LeiVMed („Leistungsvergleich Medizin“) institutionalisiert. 2017 wurde die Forschungsgruppe Bioinformatik am Campus Hagenberg in LeiVMed integriert, die zusätzliche Kompetenzen in den Bereichen Informatik und Machine Learning (ML) einbringt. LeiVMed zielt auf den Vergleich von medizinischen Behandlungen operierender Fächer mit hoher Frequenz und Potenzial zur Standardisierung der jeweiligen Behandlungsklasse (zum Beispiel Kataraktoperation) ab (vgl. [Com03]). Zurzeit arbeitet LeiVMed an einem Benchmarking-Projekt mit der Holding der elf oberösterreichischen Landeskrankenhäuser (OÖG).
Adäquates Benchmarking bedingt einerseits eine valide sowie faire Berechnung und übersichtliche Aufbereitung von Kennzahlen auf Basis eines repräsentativen Anteils an behandelten medizinischen Fällen. Andererseits bedingt es eine valide sowie zeitnahe Datenerhebung und -aufbereitung zu vernünftigen Kosten. Da die Datenaufbereitung immer wieder aufs Neue herausfordernd ist, sind die Transformation und die Plausibilitätsprüfung der aus Krankenhaus-Applikationen extrahierten Daten Gegenstand dieses Artikels. Die Berechnung und Visualisierung der Kennzahlen sind in LeiVMed mittels REST-Services und Web-Frontend voll automatisiert. In diesem Artikel werden weniger in LeiVMed angewandte Methoden zur Datenaufbereitung selbst als die Prozesse und Regeln zu ihrer Anwendung, also die Governance von LeiVMed, fokussiert (vgl. [GlC16]). Datenschutz ist in LeiVMed, angesichts der sensiblen Daten, natürlich von zentraler Relevanz, betrifft die Datenaufbereitung jedoch nur am Rande und würde den Rahmen dieses Artikels sprengen.
Ausgangssituation
Medizin-Benchmarking impliziert einerseits die Verwendung von Daten aus medizinischen Fachabteilungen verschiedener Krankenhäuser. Das wiederum impliziert in der Regel Daten verschiedener Applikationen (zum Beispiel SAP IS-H) und damit Daten in verschiedenen Formaten sowie Strukturen und unterschiedliche Stammdaten (zum Beispiel medizinische Leistungen). Andererseits sind die bei medizinischen Behandlungen anfallenden Daten wie Befunde, Arztbriefe etc. weitgehend unstrukturierte Daten (trotz Standards zum Strukturieren derartiger Daten wie zum Beispiel HL7 CDA). Davon sind auch Daten über medizinische Komplikationen (zum Beispiel Wundinfektionen) betroffen [BeG16]. Gerade Komplikationen sind für Medizin-Benchmarking sehr wichtig, weil damit weitgehend die Ergebnisqualität medizinischer Behandlungen gemessen wird. Auswertungen wie zum Beispiel Komplikationsraten können also nicht unmittelbar auf Routinedaten basierend gemacht werden.
Strukturierte Routinedaten sind in Krankenhäusern hauptsächlich administrative Daten wie etwa das Aufnahmedatum. Hier haben Krankenhäuser international auch großes Potenzial bezüglich der Datenqualität und des Managements von Stammdaten. Medizinische Behandlungsklassen sowie deren Dokumentation in Krankenhaus-Applikationen sind weitgehend nicht standardisiert [ArG17]. Neben Änderungen der Umweltbedingungen oder der Organisation von Krankenhäusern führt das zu (teilweise unangekündigten) syntaktischen oder semantischen Änderungen von Benchmarking-Daten in LeiVMed.
LeiVMed-Infrastruktur
Die Entwicklung einer Infrastruktur zur Datenaufbereitung in LeiVMed begann 2010 mit dem von der österreichischen Forschungsförderungsgesellschaft geförderten Forschungsprojekt „Ontologie-basierte Benchmarking-Infrastruktur für Krankenhäuser“ (OBIK). Seitdem wird diese Infrastruktur kontinuierlich verbessert. Die Governance zur Datenaufbereitung in LeiVMed besteht neben einer Ontologie aus Prozessen und Regeln zur Transformation sowie Prüfung von Daten, wie in Abbildung 1 dargestellt. Die validierten Daten verbessern kontinuierlich die Ontologie, die berechneten Benchmarks dienen der kontinuierlichen Verbesserung der medizinischen Behandlungsklassen selbst.
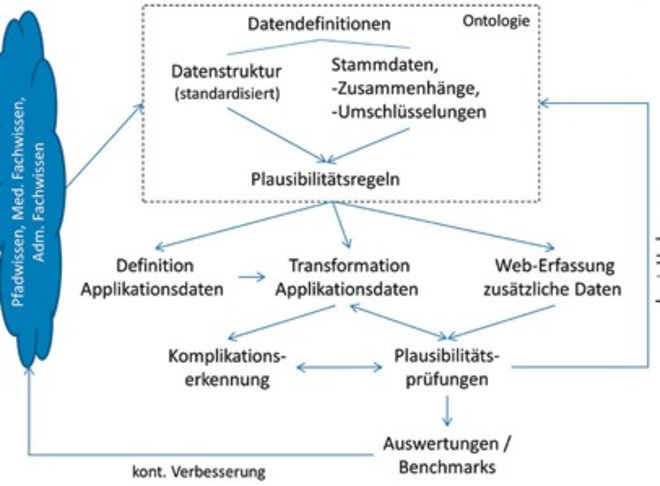
Abb. 1: Ontologie, Prozesse und Regeln zur Datenaufbereitung in LeiVMed
LeiVMed-Ontologie
Die LeiVMed-Ontologie repräsentiert, wie ebenfalls in Abbildung 1 skizziert, eine Referenz-Datenstruktur für Krankenhausinformationssysteme (KIS) sowie Stammdaten in Form von weitgehend standardisierten, medizinischen Konzepten und deren Beziehungen zueinander. Bei den verwendeten Standard-Nomenklaturen steht SNOMED CT an erster Stelle, die in der LeiVMed-Ontologie hauptsächlich medizinische Leistungen, daneben Risikofaktoren, Komplikationen sowie Laborparameter abbildet (vgl. [Sno]). Zusätzlich sind ICD10 (International Classification of Diseases, Version 10) und Stammdaten-Kataloge gemäß der österreichischen, leistungsorientierten Krankenhaus-Finanzierung (LKF) in Verwendung. Die medizinischen Konzepte der LeiVMed-Ontologie repräsentieren damit eine „einheitliche Sprache“, auf deren Basis Abteilungen von Krankenhäusern (mit weitgehend proprietären Stammdaten-Definitionen) verglichen werden können.
Die verwendeten Standard-Nomenklaturen haben in LeiVMed nur Referenz-Charakter und sind erweitert, falls zum Beispiel SNOMED keine Definition für eine relevante medizinische Leistung bietet. Die Beziehungen zwischen den Konzepten bilden Hierarchien und Kategorien von medizinischen Konzepten, Umschlüsselungen zu Stammdaten von Krankenhäusern und semantische Plausibilitätsregeln (zum Beispiel „Diagnose bzw. Komplikation Lungenentzündung verlangt nach medizinischer Leistung Lungenröntgen“) ab.
Damit ist die Ontologie die semantische Basis für die Transformation von aus KIS extrahierten Daten, einer Web-Applikation zur manuellen Ergänzung dieser Daten und zur Erfassung von Trainingsdaten für ML-Algorithmen durch medizinische Experten (Study Nurses). Außerdem ist sie eine Referenz für Datenelemente in KIS. ML-Algorithmen und damit generierte Modelle werden in LeiVMed für die (retrospektive) Erkennung von Komplikationen in medizinischen Fällen verwendet. Die aus KIS zu extrahierenden Daten entsprechen weitgehend Standard-Nachrichten (zum Beispiel Minimum Basic Data Set laut LKF), womit der Aufwand für beteiligte Krankenhäuser gering gehalten und die Qualität der extrahierten KIS-Daten gefördert wird.
Plausibilitätsprüfungen
Automatische sowie manuelle Plausibilitätsprüfungen validieren die extrahierten KIS-Daten sowie die klassifizierten Komplikationen in jedem Benchmarking-Intervall. Der zeitliche Ablauf der Daten-Transformationsschritte und der Plausibilitätsprüfungen sowie die Verantwortung dafür sind in einem Datenaufbereitungsprozess im Detail definiert. Dieser Prozess ermöglicht die exakte Koordination der Datenaufbereitung und fördert damit nicht nur die Validität, sondern auch die Aktualität der Daten und die Einhaltung der geplanten Kosten. Zusammenfassend sind in diesem Prozess folgende Plausibilitätsprüfungen koordiniert:
Zuerst werden die extrahierten KIS-Daten der syntaktischen Prüfung auf Entsprechung der Import-Spezifikation von LeiVMed unterzogen. Anschließend werden triviale semantische Prüfungen wie „Mind. 1 Operation je Kernaufenthalt vorhanden?“ durchgeführt. Daraufhin werden die Daten transformiert. Neben der Struktur-Standardisierung ist insbesondere das Transformieren von administrativen Fällen zu medizinischen Fällen und damit Daten aus medizinischer Perspektive Gegenstand dieser Transformation. Die transformierten Daten werden dann mit komplexeren Fragestellungen wie „Verantwortliche Abteilung für jeden medizinischen Fall generiert?“ oder „Behandlungsklassen passen fachlich zu verantwortlichen Abteilungen?“ geprüft. Des Weiteren wird durch statistische Analyse (Quantil-Berechnungen) die Verteilung von Parameter-Häufigkeiten (zum Beispiel Leistungsprofile medizinischer Fälle) und Parameter-Werten geprüft. Diese semantischen Prüfungen werden mittels Auswertungen wie zum Beispiel „medizinische Fälle ohne Operation“ durchgeführt.
Abschließend findet eine automatische Kontrolle der Modelle und auch ihrer Dateninputs mittels Cross Validation statt. Es werden alle medizinischen Fälle mit erkannter Komplikation sowie eine Stichprobe der medizinischen Fälle ohne erkannte Komplikation manuellen Prüfungen durch Study Nurses unterzogen. Gegebenenfalls daraufhin durchgeführte, manuelle Korrekturen der Ergebnisse führen in weiterer Folge zur Verbesserung der Modelle.
Komplikationserkennung
In LeiVMed werden medizinische Fälle als komplikativ oder nicht komplikativ erkannt bzw. klassifiziert. Komplikative Fälle werden in schwerwiegend (major) und moderat (minor) unterteilt. Auf Trainingsdaten aufbauend wurden mathematische Modelle identifiziert, welche diese Einteilung der Fälle automatisch vornehmen können. Für das Generieren dieser Modelle werden aus KIS extrahierte Daten sowie manuell erfasste Daten zu medizinischen Komplikationen verwendet. Damit können komplikative Fälle auch (retrospektiv) als solche klassifiziert werden. Für die Modellgenerierung werden verschiedene ML-Algorithmen verwendet, unter anderem symbolische Klassifikation (Abbildung 2A), künstliche Neuronale Netze (Abbildung 2B), Random Forests und Support Vector Machines.
Für die endgültige Klassifikation eines medizinischen Falles als major/minor/nicht komplikativ wird das Prinzip des Multi-Modell-Ensembles (MME) herangezogen [Win14]. Damit wird die Klassifikation der Fälle nochmals verbessert.
Durch die Plausibilitätsprüfungen in LeiVMed wird zusätzlich die Validität der Modelle laufend überprüft. Ist diese Validität nicht mehr gegeben, werden betroffene Modelle erneut trainiert. Beim Einsatz der Modelle werden in LeiVMed folgende Regeln berücksichtigt:
- Beständigkeit der verwendeten Parameter
- Unabhängige Modelle
- Diversifizierung der Algorithmen
Ein sehr wichtiger Faktor beim Trainieren und Verwenden von Modellen in LeiVMed ist die Beständigkeit der verwendeten Parameter bzw. Features. Dies schließt die eindeutige Identifikation und Beibehaltung von Features ein, die in LeiVMed für das Trainieren der Modelle verwendet werden. In geringem Maße unvollständige Daten können durch die Verwendung des MME-Ansatzes kompensiert werden, da nicht alle Modelle dieselben Features für die Klassifikation verwenden und somit nur die Anzahl der entscheidungsfähigen Modelle reduziert wird. Redundante Parameter werden automatisiert bestimmt, in weiterer Folge manuell überprüft und gegebenenfalls in der Modellierung nicht berücksichtigt.
Es wird darauf geachtet, dass unabhängige Modelle unter Verwendung verschiedener Algorithmen trainiert werden. Ebenso wichtig ist es, dass relevante Parameter, wie zum Beispiel bestimmte medizinische Leistungen, in KIS nicht entfernt oder verändert werden. Werden den Daten neue Messwerte oder sonstige Parameter hinzugefügt oder entfernt, werden neue Modelle trainiert, die mit bzw. ohne diese Features auskommen.
Schließlich wird bei systembedingten Änderungen in den Daten durch Adaptierung der einstellbaren Parameter eine Diversifizierung der Algorithmen in LeiVMed durchgeführt. Dieser kontinuierliche Verbesserungsprozess führt zu robusteren und heterogenen Sets von Modellen. Systembedingte Änderungen in den KIS-Daten können etwa durch neue medizinische Verfahren oder fortschreitende Digitalisierung in Krankenhäusern entstehen (vgl. [Scu15]).
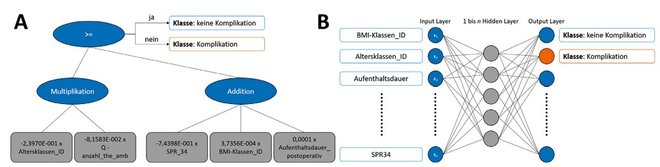
Abb. 2: Beispielhafte Darstellung zweier Modelle. Links (A) ein durch genetische Programmierung gelerntes symbolisches Klassifikationsmodell, rechts (B) ein Neuronales Netz
Praxiserfahrung
Nach ersten Auswertungen können in LeiVMed mit ML-Verfahren medizinische Fälle zu ca. 90 Prozent richtig klassifiziert werden. Insgesamt werden ca. 10 Prozent der Fälle als komplikativ eingestuft, was weit über den tatsächlichen Komplikationsraten liegt. Die Modelle sind auf das Erkennen möglichst aller tatsächlich aufgetretenen Komplikationen trainiert und klassifizieren dadurch eher zu viele als zu wenige Fälle als komplikativ, was zusätzlichen Aufwand für die Study Nurses bedeutet. Rund 90 Prozent der schwerwiegend komplikativen Fälle werden erkannt. Dabei ist zu beachten, dass die Anteile richtig klassifizierter Fälle, als komplikativ eingestufter Fälle und als schwerwiegend komplikativ erkannter Fälle nicht unmittelbar voneinander abhängig sind – die entsprechenden Zahlen schaffen den Anschein der gegenseitigen Abhängigkeit nur zufällig.
Durch den Einsatz von ML-Methoden kann LeiVMed aktuell 45.000 medizinische Fälle (von 75.000 in den untersuchten Abteilungen insgesamt behandelten) pro Jahr (bei monatlicher Publikation der Benchmarks) mit 1,5 Vollzeit-Äquivalenten für Datenaufbereitung und -validierung bewerten und vergleichen. Hohe Flexibilität und eine relativ geringe Komplexität der in der Datenaufbereitung verwendeten Methoden und Prozeduren ist ebenfalls gegeben. Governance ist für LeiVMed von zentraler Relevanz: Ohne adäquate Governance wäre die Datenaufbereitung insbesondere mit dem Einsatz von ML-Methoden schwer realisierbar.
Fazit
Die LeiVMed-Infrastruktur ermöglicht zeitnahes Benchmarking für rund 60 Prozent der medizinischen Fälle von operierenden Abteilungen auf der Basis von Routinedaten und zu vernünftigen Kosten. Durch die manuelle Nachkontrolle der Study Nurses kann die Rate der korrekt klassifizierten medizinischen Fälle auf über 90 Prozent gehoben werden. Damit können Krankenhausmanager neue Einblicke in operierende Abteilungen ihrer Spitäler gewinnen und fundierte Maßnahmen zur deutlichen Steigerung der Qualität von medizinischen Behandlungen bei gleichzeitiger Senkung der Kosten ergreifen.
Weitere Informationen
[ArG17]
Arthofer, K. / Girardi, D.: Data quality- and master data management, a hospital case. In: Stud Health Technol Inform. Health Informatics meets eHealth, 2017, S. 259–266
[BeG16]
Benson, T. / Grieve, G.: Principles of health Interoperability. Springer 2016, S. 19–32
[Com03]
Institute of Medicine: Leadership by Example: Coordinating Government Roles in Improving Health Care Quality. National Academy Press 2003
[GlC16]
Gluchowski, P. / Chamoni, P.: Analytische Informationssysteme. Business-Intelligence-Technologien und -Anwendungen. Springer Gabler 2016
[Scu15]
Sculley, D. et al.: Hidden Technical Debt in Machine Learning Systems. NIPS 2015
[Sno20]
SNOMED International: 5-step Briefing. Aktualisiert 31.1.2020,
www.snomed.org/snomed-ct/five-step-briefing, abgerufen am 30.7.2020
[Win14]
Winkler, S. M. et al.: Data Based Prediction of Cancer Diagnoses Using Heterogeneous Model Ensembles – A Case Study for Breast Cancer, Melanoma, and Cancer in the Respiratory System. Proceedings of the Genetic and Evolutionary Computation Conference GECCO 2014